若從野外抓30隻公鳥測量體重,公鳥bird變項=1,其中一隻叫「阿財」,故此群組簡稱「阿財及其兄弟們」。
 |
| 我叫阿財。 |
若再從同區域野外抓18隻母鳥測量體重,母鳥bird變項=0,其中一隻叫「灰皮」,故此群組簡稱「灰皮及其姐妹們」。
 |
| 我叫灰皮醬,小名叫灰皮。 |
接下來,將利用SAS簡易比較高雄市三民區野外玄鳯鸚鵡平均體重於公母鳥群體間是否存在顯著統計差異,並將比較結果繪製成統計圖。
data bird;
input bird weight; /*依序灌入2個群組所屬鳥類的體重*/
datalines;
1 84
1 83
1 84
1 80
1 81
1 95
1 92
1 92
1 92
1 95
1 95
1 92
1 95
1 96
1 98
1 103
1 99
1 93
1 88
1 88
1 88
1 98
1 101
1 100
1 100
1 104
1 98
1 100
1 99
1 95
0 102
0 107
0 100
0 104
0 95
0 111
0 115
0 112
0 103
0 95
0 103
0 120
0 115
0 115
0 110
0 110
0 109
0 98
;run;
我的資料合條件「總樣本數≥ 30且每一群組樣本≥ 10」。
再評估有否符合「數列呈常態分佈」的假設。
proc univariate data=bird plot normal;
var weight;
where bird=1;
run;
結果如下:
出處:https://www.stat.purdue.edu/~tqin/system101/method/QQplot_sas.htm
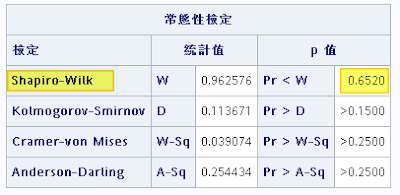
p=0.1134>0.05, 故該群組數列呈常態分布,以上是客觀判斷是否為常態分布方法;以下是主觀判斷方法
若真的只想判定有無符合常態分布,只想輸出上述的常態性檢定表,可在proc univariate中,加入Ods select testsfornormality; 即可達成。
左上圖分布是否呈鐘形曲線、右上圖中位數線與平均數icon會不會很接近、下圖的資料點分佈是否延著該直線分布。若上述答案為是,則表示為常態分布。
再評估另一群組是否也符合「數列呈常態分佈」的假設。
proc univariate data=bird plot normal;
var weight;
where bird=0;
run;
結果如下:
故兩群組均符合常態分布。
再來評估有否符合「2群組具有相似的變異數(變異數同質性)」的假設。
proc format; /*設定自建的格式,格式名稱叫birdfmt*/
value birdfmt 1='阿財及其兄弟們' 0='灰皮及其姐妹們' ;run;
proc ttest data=bird cochran;
class bird;
var weight;
format bird birdfmt.; /*將bird變項套用自建的格式birdfmt*/
run;
結果如下:
p=0.5961 >0.05, 表示兩群組間的變異數具同質性,合假設。
故本案例已符合1個條件,2個假設,所以可以student t test評估平均數差異性。
上面即是t test評估的結果,該程式預設輸出圖表尚包含如下(版大未列全部):
本例採用集區的 Pr >|t| ,p<0.0001,表示體重平均數在兩群組間,呈統計學上顯著差異。
即可推論高雄市三民區野外玄鳯鸚鵡平均體重於公母鳥間呈統計上顯著差異。
若未符合條件或假設1,改用Mann-Whitney U test
若僅未符合假設2,改用Satterthwaite test 或 Cochran t-test
上述的集區,正式名稱為Pooled t-test
上述的Satterthwaite test 即在SPSS報表中,出現於下圖左下處的「不採用相等變異數」,該評估方式就是用Satterthwaite test算出 (其內的數字與本案無關,此僅舉例)。
上下兩圖已很清楚,不再說明。
若要設定統計圖長寬大小,請使用ods graphics / width= ??? px height=??? px ;
及ods graphics off;
將 proc ttest 程序上下夾起來,如下。
上述提到「可推論高雄市三民區野外玄鳯鸚鵡平均體重於公母鳥間呈統計上顯著差異。」
但你相信嗎?嘿嘿...
版大來算統計檢定力
母體真的存在差異,而抽樣評估時也存在差異的機率為1-beta,即統計檢定力power。
ods noproctitle; (以下暫不詳細說明)
ods output output=work.stat;
proc power;
TwoSampleMeans test=diff
Alpha = 0.05
Sides = 2
GroupMeans = (106.9 93.6)
StdDev = 9.3918
Power = .
GroupNs = (18 30)
;run;
即母群體若有差異,我們的統計考驗在此研究設計及樣本大小下,會有99.6%機率得到顯著差異;反之,只有0.4%的機率得到未顯著的差異。在大部份的研究領域,power設定0.8即可,但若屬臨床新藥試驗研究,通常會被期刊要求至少0.9。
故當我下「高雄市三民區野外玄鳯鸚鵡平均體重於公母鳥間呈統計上顯著差異。」結論時,我可以下的非常有信心。
過來,版大示範漂亮的盒形圖及點散布圖疊圖。(以下暫不詳細說明)
title bcolor=cream height=1.8 "圖1 寵物鳥體重盒形圖及散布圖疊圖" ;
ods graphics / imagefmt=png width= 800px height=500px ;
proc sgplot data=bird noautolegend;
hbox weight / category=bird boxwidth=0.8 boxwidth=0.5 nofill lineattrs=(thickness=2.8)
medianattrs=(thickness=2.8) meanattrs=(size=16 symbol=star) whiskerattrs=
(thickness=2.8) outlierattrs=(size=12) ;
scatter x=weight y=bird / jitter filledoutlinedmarkers markerattrs=(symbol=circlefilled size=14)
markerfillattrs=graphdata1 dataskin=sheen;
xaxis display=(nolabel) discreteorder=data;
xaxis offsetmax=0.1 label='體重(公克)' labelattrs=(size=12) valueattrs=(size=12);
yaxis display=(nolabel) valueattrs=(size=12) ;
format bird birdfmt.;
run; title;
ods graphics off;
title '圖2 寵物鳥體重群組直方圖';
proc univariate data=bird noprint;
class bird ;
var weight;
histogram weight / vscale=count vaxislabel='次數' cfill=cx80bf1a normal(noprint) barlabel=count;
label weight='寵物鳥體重';
inset normal(mu sigma);
format bird birdfmt.;
run;title;















沒有留言:
張貼留言